Une performance maximale de 1,01 exaFLOPS, selon des estimations prudentes.
Comme des millions d’utilisateurs, vous avez peut-être mis votre PC à contribution pour aider à faire avancer la recherche sur le coronavirus via Folding@Home. Un article publié dans la revue Nature revient sur cette mobilisation et ses effets.
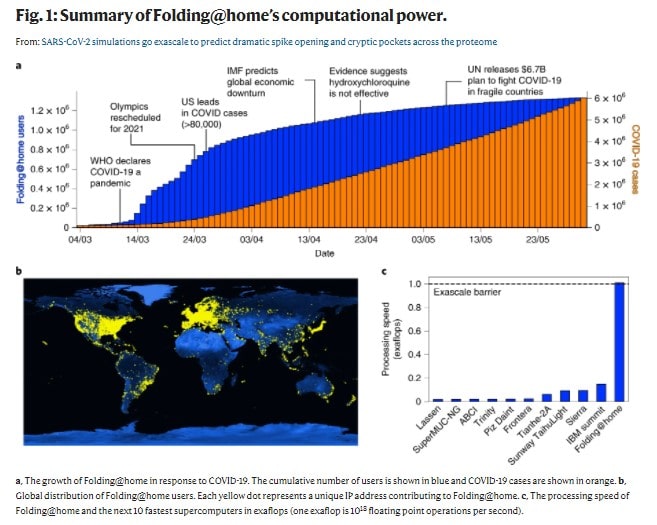
Il rapporte qu’en moins de trois mois, le nombre de machines actives à travers le globe est passé de 30 000 à plus d’un million. Rapidement, la puissance de calcul obtenue par ce biais a dépassé celles de plusieurs des plus puissants supercalculateurs réunis ; elle a franchi la barre de l’exaFLOPS. Si en avril, le compte Twitter officiel de Folding@home annonçait une puissance totale de 2,4 ExaFLOPS, pour le Coronavirus Sars-CoV-2, les auteurs de l’étude dont il est question ici estiment “prudemment que la performance maximale de Folding@home a atteint 1,01 exaFLOPS. Cette performance a été atteinte à un moment où environ 280 000 GPU et 4,8 millions de cœurs de CPU effectuaient des simulations. Comme nous l’expliquons dans la section Méthodes, pour être prudents quant à nos affirmations, nous supposons que chaque GPU/CPU a des performances inférieures à celles d’une d’une puce sortie avant 2015”.
Quoi qu’il en soit, même 1,01 exaFLOPS reste “cinq fois plus élevée que la performance maximale du superordinateur traditionnel le plus rapide du monde à l’époque, Summit. Cette performance surpasse celle des 100 premiers superordinateurs réunis. Avant Folding@home, le premier supercalculateur exascale ne devait pas être mis en ligne avant la fin de 2021.”
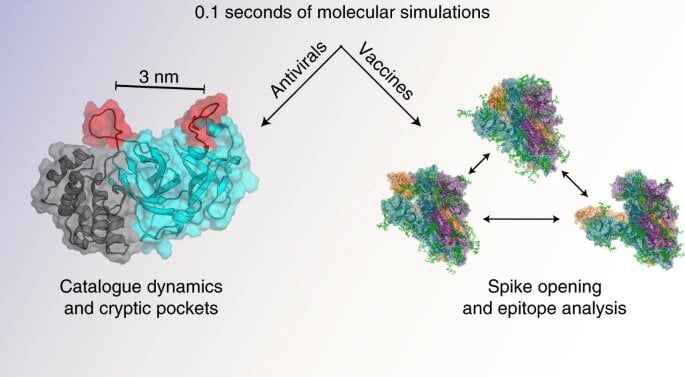
0,1 seconde de simulation
En pratique, cela a permis de simuler 0,1 seconde du protéome du Coronavirus Sars-CoV-2 : “En utilisant cette ressource, nous avons construit des cartes quantitatives des ensembles structurels de plus de deux douzaines de protéines et de complexes relatifs au SRAS-CoV-2 à partir de millisecondes de données de simulation générées pour chaque système. Ensemble, nous avons effectué 0,1s de simulation”. Toujours selon l’article, les découvertes réalisées “élargissent les options de ciblage pour la conception d’antiviraux”. Enfin, les auteurs précisent que “toutes les données et tous les modèles sont disponibles gratuitement en ligne, fournissant ainsi un atlas structurel quantitatif”.
Source : Nature













Folding@Home, c’est aussi gourmand que miner de la cryptomonnaie mais tellement plus utile à l’Humanité.